宏基因组按:NCBI物种分类注释信息格式复杂,存在层级不整齐、缺失、名称变动等问题,在使用中存在一定困难。最近发现了一款分类信息查询和格式化的工具TaxonKit,该工具于2019年发布于Biorxiv上,目前已经被引用19次(Google学术,截止2020年2月7日)。推荐给大家,助力NCBI分类信息的使用和宏基因组数据分析。

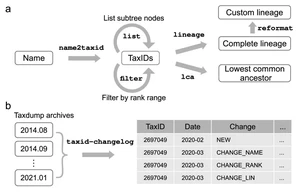
图形摘要:软件的主要功能。a. 分类信息名称-ID转换、过滤、查询、LCA、格式化等。b. 名称历史追溯。
从事生物多样性的研究者对NCBI Taxonomy数据库一定不会陌生,
它包含了NCBI所有核酸和蛋白序列数据库中每条序列对应的物种名称与分类学信息。
大多数生态学研究对物种组成的描述都是基于NCBI Taxonomy数据库,
当然目前也开始使用其他数据库,如GTDB等。
NCBI Taxonomy数据库始于1991年,一直随着Entrez数据库和其他数据库更新,
1996年推出网页版。NCBI Taxonomy数据库官方地址为 https://www.ncbi.nlm.nih.gov/taxonomy ,
公开数据下载地址为 https://ftp.ncbi.nih.gov/pub/taxonomy/ ,
数据每小时更新,每个月初生成一份数据归档存于 taxdump_archive 目录,最早可追溯到2014年8月。
TaxonKit是采用Go语言编写的命令行工具,
提供Linux, Windows, macOS操作系统不同架构(x86-64/arm64)的静态编译的可执行二进制文件。
发布的压缩包不足3Mb,除了Github托管外,还提供国内镜像供下载,同时还支持conda和homebrew安装。
用户只需要下载、解压,开箱即用,无需配置,仅需下载解压NCBI Taxonomy数据文件解压到指定目录即可。
- 源代码 https://github.com/shenwei356/taxonkit ,
- 文档 http://bioinf.shenwei.me/taxonkit (介绍、使用说明、例子、教程)
选择系统对应的版本下载最新版 https://github.com/shenwei356/taxonkit/releases ,解压后添加环境变量即可使用。或可选conda安装
测试数据下载可直接 https://github.com/shenwei356/taxonkit 下载项目压缩包,或使用git clone下载项目文件夹,其中的example为测试数据
TaxonKit为命令行工具,采用子命令的方式来执行不同功能,
大多数子命令支持标准输入/输出,便于使用命令行管道进行流水作业,
轻松整合进分析流程中。
备注:
- 输出:
-
- 所有命令默认输出到标准输出(stdout),可通过重定向()写入文件。
- 或通过全局参数或指定输出文件,且可自动识别输出文件后缀()输出gzip格式。
- 输入:
-
- 输入格式为单列,或者制表符分隔的格式,输入数据所在列用或指定。
均可从标准输入(stdin)读取输入数据,也可通过位置参数(positional arguments)输入,即命令后面不带
任何flag的参数,如
list 列出指定taxID所在子树的所有TaxID
如,
list使用最广泛的的功能是获取某个类别(比如细菌、病毒、某个属等)下所有的TaxID,
用来从NCBI nt/nr中获取对应的核酸/蛋白序列,从而搭建特异性的BLAST数据库。
官网提供了相应的详细步骤:http://bioinf.shenwei.me/taxonkit/tutorial 。
lineage 根据TaxID获取完整谱系
列出lineage每个分类学单元的的taxID和rank和名称,比如SARS-COV-2。
reformat 生成标准层级物种注释
有时候,我们并不需要完整的分类学谱系(full lineage),因为很多级别即不常用,而且不完整。通常只想保留界门纲目科属种。
值得注意的是,不是所有物种都有完整的界门纲目科属种水平,特别是病毒以及一些环境样品。
TaxonKit可以用自定义内容替代缺失的分类单元,如用“__”替代。
更厉害有用的是,TaxonKit还可以用更高层级的分类单元信息来补齐缺失的层级 (),比如
输出格式可选只输出部分分类学水平,还支持制表符(),再配合作者的另一个工具csvtk,可以输出漂亮的结果。
其它有用的选项:
- :
给每个分类学水平添加前缀,比如。
- :
输出分类学单元对应的TaxID。
- : 替代没有对应rank的taxon名称
- : 对于低于species且rank既不是subspecies也不是stain的taxid,使用水平最低taxon名称做为菌株名称。
例,
name2taxid 将分类单元名称转化为TaxID
将分类单元名称转化为TaxID非常容易理解,唯一要注意的是某些taxID对应相同的名称,比如
获取TaxID之后,可以立即传给taxonkit进行后续操作,但要注意用指定taxID所在列。
filter 按分类学水平范围过滤TaxIDs
filter可以按分类学水平范围过滤TaxIDs,注意,不仅仅是特定的Rank,而是一个范围。
比如genus及以下的分类学水平,用,类似于 。
lca 计算最低公共祖先(LCA)
比如人属的例子
TaxID的分隔符可用指定,默认为” “。
除了简单的增加、删除、合并之外,作者将TaxID改变做了细分。输出格式如下
例2 SARS-CoV-2 。可见新冠病毒在2020年2月加入,随后3月和6月份改了名称,谱系等信息。查询速度也很快。
更多有意思的发现详见
https://github.com/shenwei356/taxid-changelog
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘

 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读
版权声明:
本文来源网络,所有图片文章版权属于原作者,如有侵权,联系删除。
本文网址:https://www.bianchenghao6.com/java-jiao-cheng/8298.html
